determinar la unión exógena de los patógenos al genoma del huésped en todo el genoma?
yahoo301503
Leí este documento donde modifican específicamente una región en el genoma del arroz para eliminar el sitio de unión de un patógeno, Xanthomonas oryzae , e interrumpir el secuestro de una red de genes en el genoma del arroz en beneficio del patógeno.
Es un concepto interesante, pero me dejó preguntándome cómo se determinaría de novo la unión de estos efectores patógenos en el huésped si no se los conociera. ¿Cuáles serían los pasos para determinar dónde se unen los TF del patógeno al genoma del huésped?
Editar: existe la posibilidad de no saber qué proteínas se unen al genoma del huésped, entonces, ¿cuál sería la forma de desarrollar los anticuerpos en este caso?
Referencia:
Respuestas (2)
Konrad Rodolfo
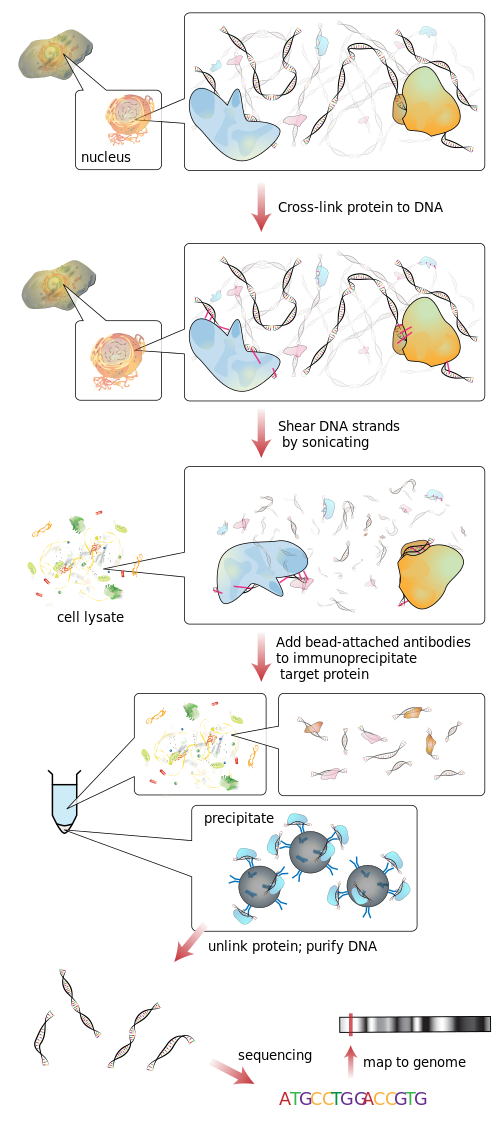
Las técnicas utilizadas para hacer esto son ChIP-seq y ChIP-chip.
Básicamente, tú
- dejar que el patógeno se una al ADN (altamente replicado)
- cortar el ADN en pequeñas piezas aleatorias mediante sonicación
- enriquecer ("tirar hacia abajo") los fragmentos de ADN unidos al patógeno mediante el uso de un anticuerpo conocido que se une al patógeno
- secuenciar el ADN así enriquecido
- mapear los fragmentos secuenciados de nuevo a su ubicación original en el genoma.
Wikipedia tiene una buena imagen que explica este proceso con un poco más de detalle:
Daniel Standage
Un colega mío descubrió el cifrado que determina las especificidades del ADN del efector TAL, que se describe en este breve artículo . Estas especificidades se determinaron observando los efectores TAL unidos al ADN y registrando la frecuencia con la que un diresiduo variable repetido (RVD) dado correspondería a un nucleótido dado (usando una matriz de peso).
Ahora que se han determinado las especificidades, identificar los sitios diana del efector TAL es simplemente una cuestión de medir la probabilidad de que la secuencia RVD del efector TAL se alinee con una secuencia nueva. Algunas aplicaciones se describen aquí y aquí .
El servidor TALENT 2.0 (artículo invitado en revisión) proporciona herramientas para diseñar efectores TAL personalizados para apuntar a secuencias de ADN específicas, o para identificar posibles sitios de unión fuera del objetivo en una secuencia o genoma dado.
Referencias:
¿Cómo se pueden determinar los sitios de unión del promotor?
¿Cuánto influye en la transcripción la distancia entre un sitio de unión del factor de transcripción y un promotor?
identificar qué SNP se encuentran en TFBS (levadura)
¿Qué hace que las secuencias de ADN sean más diferentes/reconocibles desde una perspectiva biológica? [duplicar]
Encontrar elementos regulatorios: ¿Cómo ha estado haciendo esto la gente?
Ubicación de TFBS en el genoma
Número de genes de factores de transcripción en el genoma humano
¿A qué otros sitios se unen los inhibidores no competitivos aparte de los sitios alostéricos?
Todas las células somáticas contienen el mismo genoma, entonces, ¿cómo sabe que debe convertirse en un órgano específico?
Silenciamiento génico: ¿número relativo o absoluto de sitios CpG metilados?